【Adv. Sci.】西北农林科技大学马闯:深度学习助力基因调控研究——跨物种预测转录因子结合位点的新进展
基因表达的复杂调控是生命科学中的一个核心问题。转录因子(TFs)通过识别特定的DNA序列——转录因子结合位点(TFBSs)来调控基因表达。准确预测 TFBSs对于理解基因调控网络至关重要。然而,传统的实验方法耗时且成本高昂,而现有的计算方法在数据量有限或跨物种预测时表现不佳。

2025年5月24日,西北农林科技大学马闯的研究团队开发了一个基于深度学习的框架deepTFBS,能够利用大规模TF结合数据提升预测能力,相关研究发表于《Advanced Science》。该研究不仅在拟南芥中验证了模型的高效性,还在小麦中展示了其跨物种预测的潜力。
关键信息或发现
deepTFBS 框架设计
deepTFBS 结合了多任务学习和迁移学习的优势,能够同时学习多个转录因子的结合偏好,并利用预训练模型在数据量有限的情况下进行微调。该框架的核心是一个混合神经网络结构,包括卷积层、双向长短期记忆(BiLSTM)层和单头注意力(SHA)层。这些组件协同工作,能够捕捉DNA序列中的局部和全局特征,从而提高预测精度。
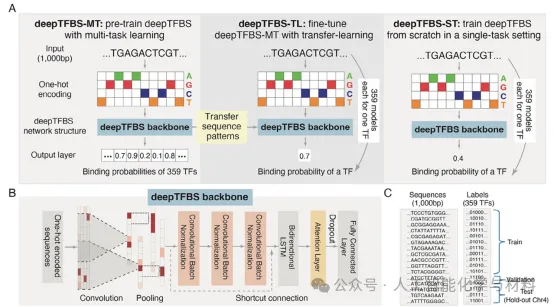
图1:deepTFBS框架示意图(图源自Adv. Sci.)
概述:该图展示了deepTFBS的三种变体:deepTFBS-MT(多任务学习)、deepTFBS-TL(迁移学习)和 deepTFBS-ST(单任务学习)。模型输入 1000 个碱基对的 DNA 序列,通过深度学习网络结构预测每个转录因子的结合概率。deepTFBS-MT 同时预测359种转录因子的结合概率,而 deepTFBS-TL 则针对特定转录因子进行微调,以提高预测精度。
性能提升
在拟南芥的359种转录因子测试中,deepTFBS的表现超越了传统的位置权重矩阵(PWM)方法、DeepSEA 和 DanQ 等现有方法。与 PWM 相比,deepTFBS 的精确率-召回率曲线下面积(PRAUC)提高了 244.49%,与 DeepSEA 和 DanQ 相比,分别提高了 49.15% 和 23.32%。在小麦的跨物种预测中,deepTFBS 的 PRAUC 提高了 30.6%,显示出其在跨物种预测中的巨大潜力。
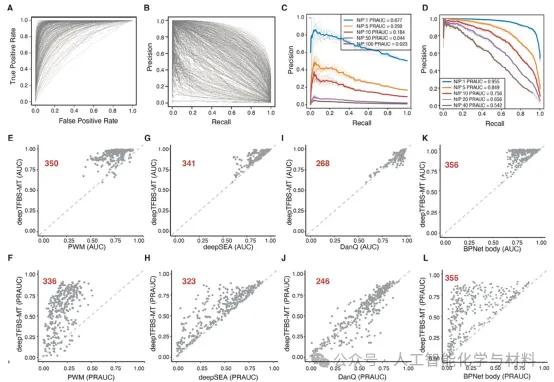
图2:deepTFBS-MT 在拟南芥中的性能评估(图源自Adv. Sci.)
概述:该图展示了 deepTFBS-MT 在预测拟南芥 359 种转录因子结合位点时的性能。通过接收者操作特征曲线(ROC)和精确率-召回率曲线(PR),deepTFBS-MT 显示出比传统方法更高的预测精度。特别是对于那些结合位点数据稀少的转录因子,deepTFBS-MT 依然能够提供可靠的预测。
模型解释能力
deepTFBS 不仅预测准确,还具有解释能力。通过集成梯度方法,研究团队能够可视化模型识别的TF结合偏好,揭示了与已知TF结合模式高度一致的序列特征。这表明 deepTFBS 能够学习并识别不同转录因子的结合语法。
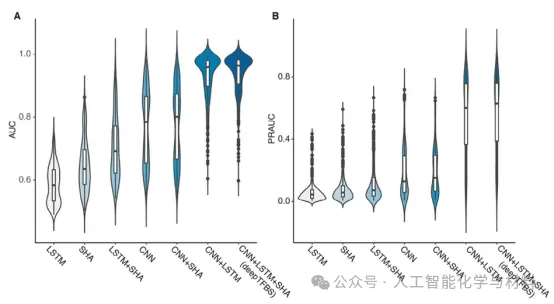
图3:deepTFBS-MT 的模型解释能力(图源自Adv. Sci.)
概述:该图通过集成梯度方法展示了 deepTFBS-MT 识别的转录因子结合偏好。通过可视化每个位置的梯度值,研究团队能够识别出对预测结果贡献最大的DNA 序列特征,从而揭示转录因子的结合模式。
功能性调控变异预测
deepTFBS 还能够预测可能影响基因表达的调控变异。通过对 1001 基因组计划中的 10706842个单核苷酸多态性(SNPs)进行分析,deepTFBS 能够量化每个 SNP 对 TF 结合的影响。研究发现,高影响 SNP 在进化上更保守,且更倾向于位于非编码序列中,这表明这些 SNP 可能在基因调控中发挥重要作用。
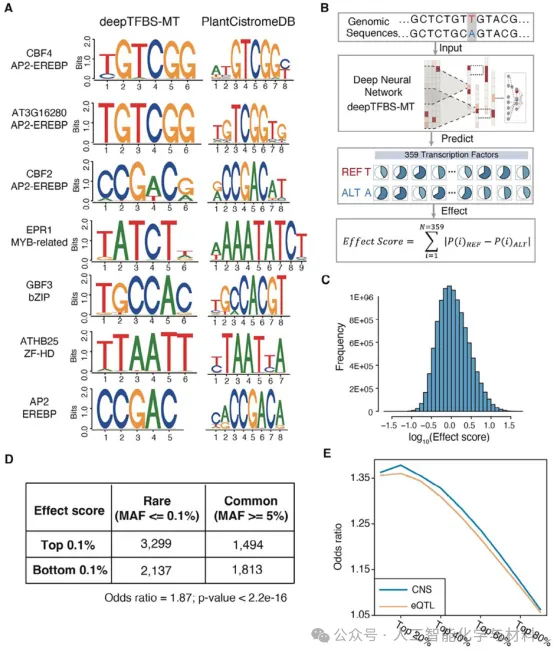
图4:deepTFBS-MT 预测的功能性调控变异(图源自Adv. Sci.)
概述:该图展示了deepTFBS-MT如何通过比较参考和替代等位基因的结合概率来预测功能性调控变异。通过计算每个SNP的影响分数,deepTFBS-MT 能够识别出可能影响基因表达的变异。研究发现,高影响 SNP 更倾向于位于进化上保守的非编码序列中。
跨物种预测
deepTFBS在跨物种预测方面表现出色。研究团队利用拟南芥预训练的模型,在小麦中进行了微调,并成功预测了小麦中的 TFBSs。这一成果不仅证明了 deepTFBS 在跨物种预测中的有效性,还为在数据稀缺的物种中应用该模型提供了可能。
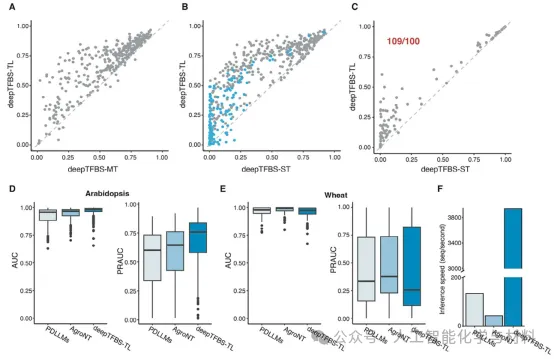
图5:deepTFBS-TL 的跨物种预测性能(图源自Adv. Sci.)
概述:该图比较了 deepTFBS-TL 和其他方法在小麦中的跨物种预测性能。结果显示,deepTFBS-TL 在预测小麦中的转录因子结合位点时表现优于其他方法,尤其是在数据量有限的情况下。这表明迁移学习能够有效提高跨物种预测的准确性。
总结与展望
deepTFBS 不仅提高了TFBSs的预测精度,还通过迁移学习和多任务学习,解决了数据量有限和跨物种预测的挑战,为基因调控研究提供了一个强大的新工具。该框架的解释能力使其能够揭示 TF 结合的潜在机制,而其在调控变异预测中的应用则为理解基因调控的遗传基础提供了新的启发。随着基因组学数据的不断增加,deepTFBS 有望在那些实验数据有限的植物基因调控网络的研究中发挥重要作用。
文章链接
https://doi.org/10.1002/advs.202503135
转载自公众号: 人工智能化学与材料